- In:
- Posted By: Capuano Edoardo
- Commenti: 0
Creato schema elettrico del sistema nervoso di un animale
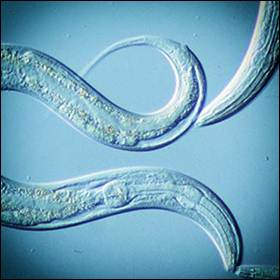
I ricercatori del College of Medicine Albert Einstein descrivono il primo schema elettrico completo del sistema nervoso di un animale, il verme Caenorhabditis elegans, usato dagli scienziati di tutto il mondo come un organismo modello. Lo studio comprende adulti di entrambi i sessi e rivela differenze sostanziali tra loro.
I risultati segnano un'importante pietra miliare nel campo della Connectomics, (1) lo sforzo di mappare le innumerevoli connessioni neurali in un cervello, nella regione dell'encefalo o nel sistema nervoso per trovare le specifiche connessioni nervose responsabili di determinati comportamenti.
Il dottor Scott W. Emmons, Ph.D., (2) leader dello studio, professore di genetica presso il Dominick P. Purpura Department of Neuroscience e la Siegfried Ullmann Chair in Molecular Genetics at Einstein, spiega: “la struttura è sempre centrale in biologia. La configurazione del DNA ha rivelato come funzionano i geni mentre la struttura delle proteine ha rivelato come funzionano gli enzimi. Ora, la struttura del sistema nervoso sta rivelando: come si comportano gli animali; come le connessioni neurali, quando sono alterate, possono causare una malattia.”


